shiny-blupADC 1
🙀大家好,从本节开始,我将给大家分享 blupADC-online (在线版)的使用说明。可能有同学会问:既然已经有了blupADC 这个package了,那么为什么还要开发blupADC-online 呢?
其实,blupADC-online可以看成是blupADC package的简化版。凡是blupADC-online 能实现的功能, blupADCpackage均能实现。但是,考虑到部分用户可能对R不太了解,因此我们想到了开发blupADC-online 。通过blupADC-online ,用户即使没有编程基础也能很快的完成相应的分析💯。
本节,我们给大家带来的是如何通过blupADC-online 完成DMU相关的分析。
✋登录
在浏览器栏直接输入网址: http://47.95.251.15:443/sample-apps/blupADC/ 即可登录到blupADC-online主界面,如下所示:
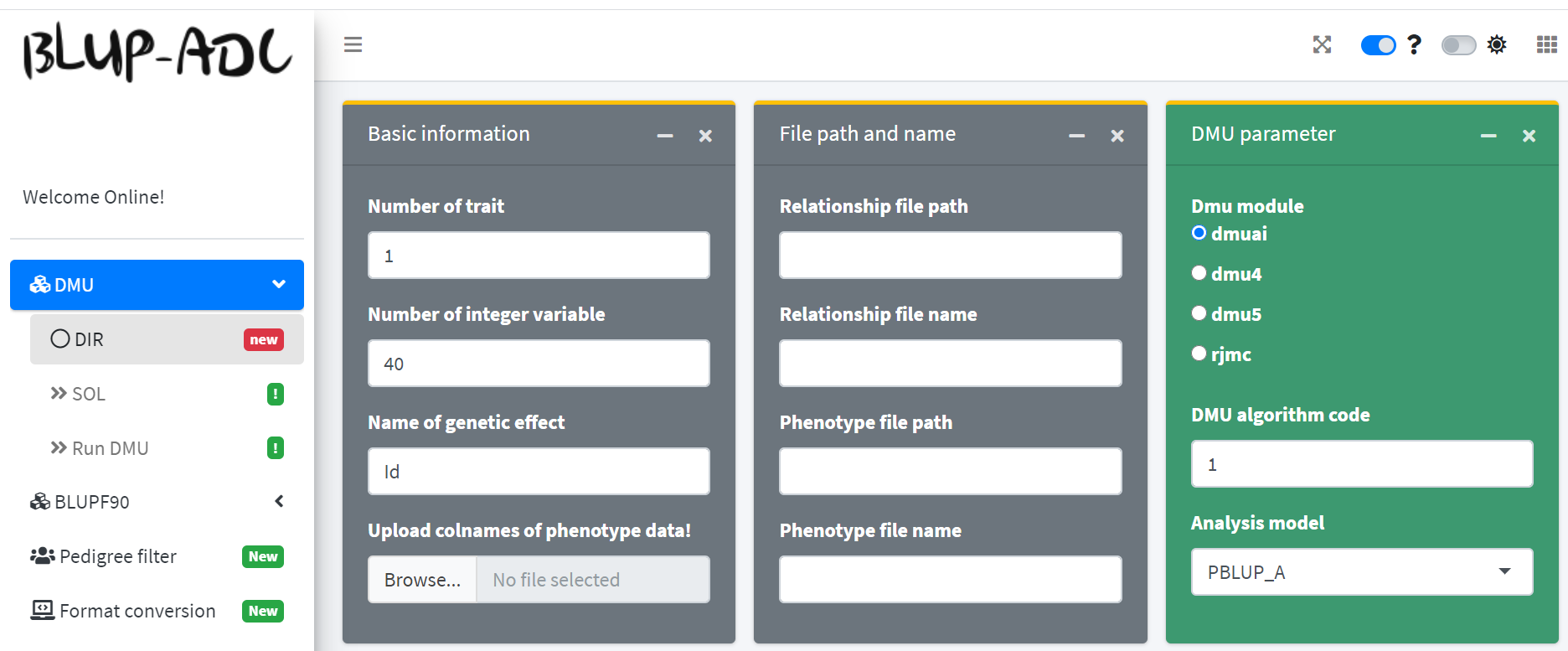
首先我们在菜单栏点击DMU图标,即可发现有三个子菜单,从上到下依次是:
DIR、SOL 和 Run DMU。本节的重点就是介绍这三个子菜单界面。
如果大家事先已经了解过run_DMU 函数的话,那么本节内容可能对大家来说就比较熟悉了。如果没有事先了解也没关系,因为相关的操作还是非常简单的。
✋DIR界面
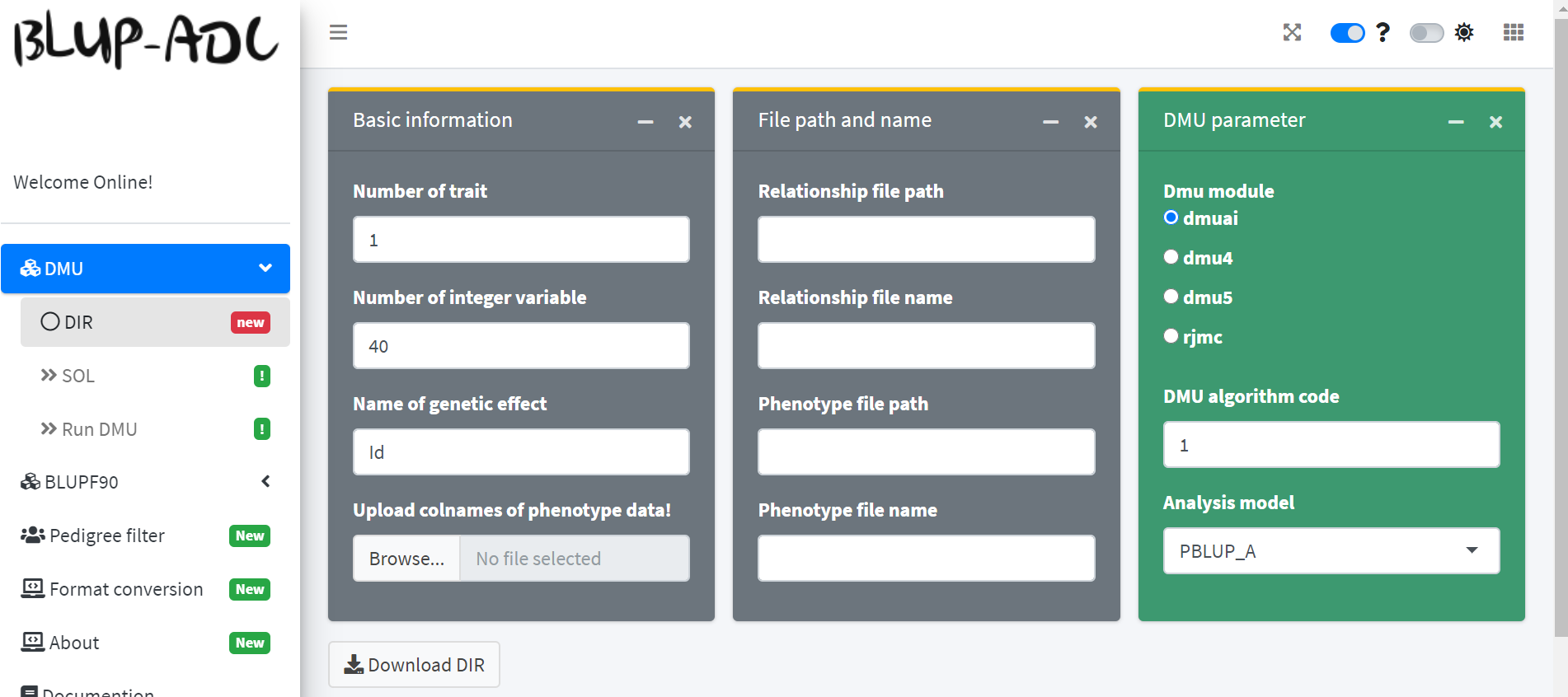
点击DIR菜单,我们就能进入 DIR界面。DIR界面的主要功能是生成 DMU格式的参数文件。下面我们来对界面内的各个参数进行逐一讲解:
Basic information
Number of trait: 要分析的性状的数目Number of integer variable: 表型数据中,整型变量的个数Name of genetic effect: 遗传效应的名称(一般为动植物个体Id)Upload colnames of phenotype data: 上传表型数据的列名文件。文件格式如下所示:
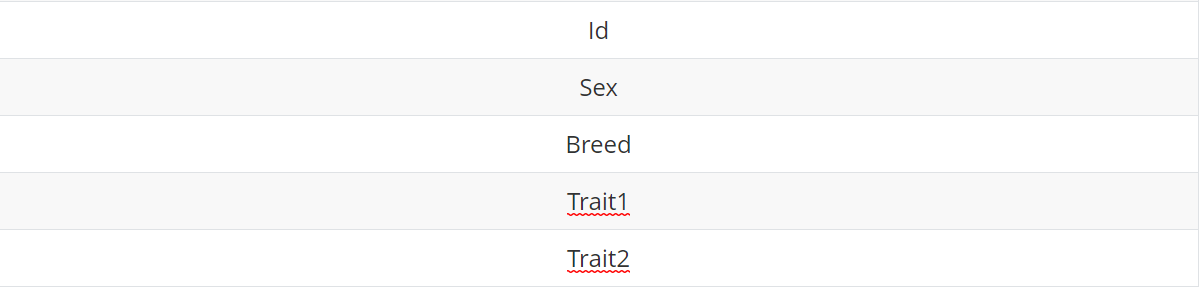
Note: 列名文件的格式为:n行1列。每行代表的是表型数据的列名。
File path and name
Relationship file path: 亲缘关系文件的路径Relationship file name: 亲缘关系文件的名称。针对不同的Analysis model,我们需要提供不同的亲缘关系文件。具体对应关系为:
- PBLUP_A: 亲缘关系文件为系谱文件
- GBLUP_A: 亲缘关系文件为基因组亲缘关系矩阵的逆矩阵文件
- GBLUP_AD: 亲缘关系文件为基因组亲缘关系矩阵的逆矩阵文件
- SSBLUP_A: 亲缘关系文件为系谱文件及基因组亲缘关系矩阵文件
Phenotype file path: 表型文件的路径Phenotype file name: 表型文件的名称
DMU parameter
DMU module: DMU进行遗传评估时所选用的模块,默认为 dmuai 。DMU algorithm code: 方差组分估计所用的算法,默认为 1。Analysis model: 进行遗传评估所选用的模型。可选模型包括:- PBLUP_A: 系谱加性效应模型
- GBLUP_A: 基因组加性效应模型
- GBLUP_AD:基因组加性-显性效应模型
- SSBLUP_A:一步法加性效应模型
当用户上传表型数据的列名文件后,当前界面内便会新增几个窗口,如下:
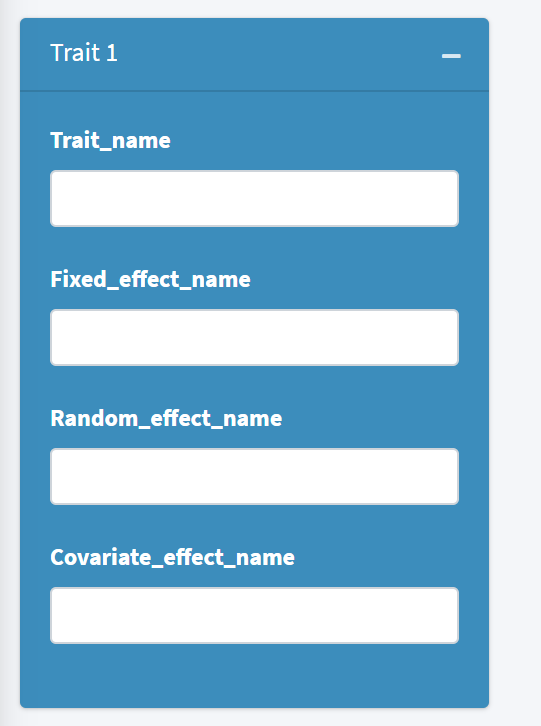
用户可以通过设置Number of trait来调整小窗口的个数。
e.g. 当设置Number of trait为3时,就会生成3个小窗口,如下所示:
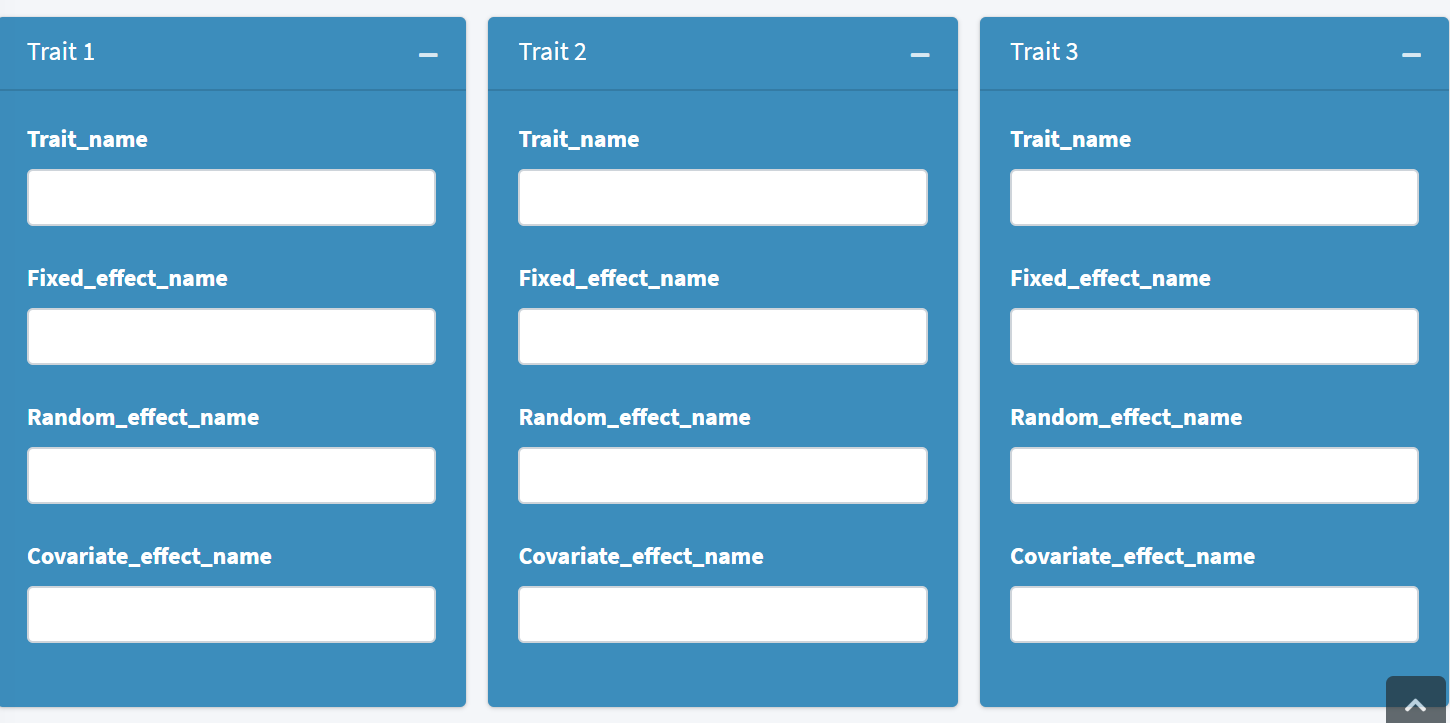
用户可以在窗口内输入想要分析的性状名称及对应的固定效应、随机效应和协变量效应的名称。为了方便用户输入,选择性状和效应名称时可通过下拉菜单选取,如下:
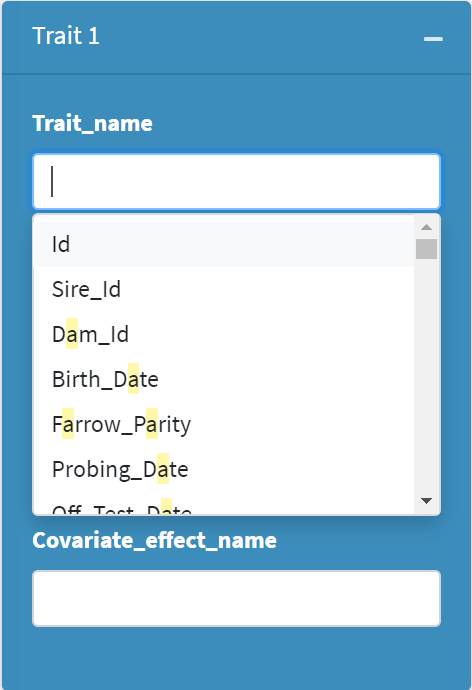
下面以一个具体的例子进行说明:
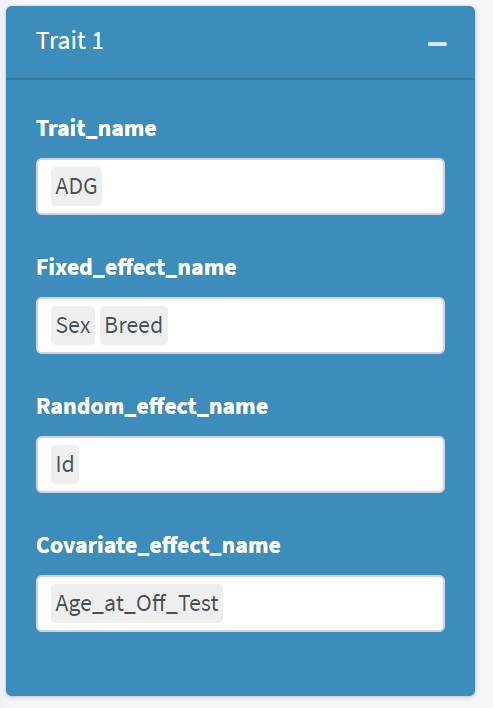
e.g. 想要分析的性状为:ADG ;
固定效应包括: Sex 和 Breed;
随机效应包括Id ;
协变量效应包括:Age_at_off_test。
那么我们只需要在窗口内输入对应的信息即可,如下:
输入完相应的效应后,在界面的正下方会实时显示所生成的 dir 参数文件内容,如下所示:
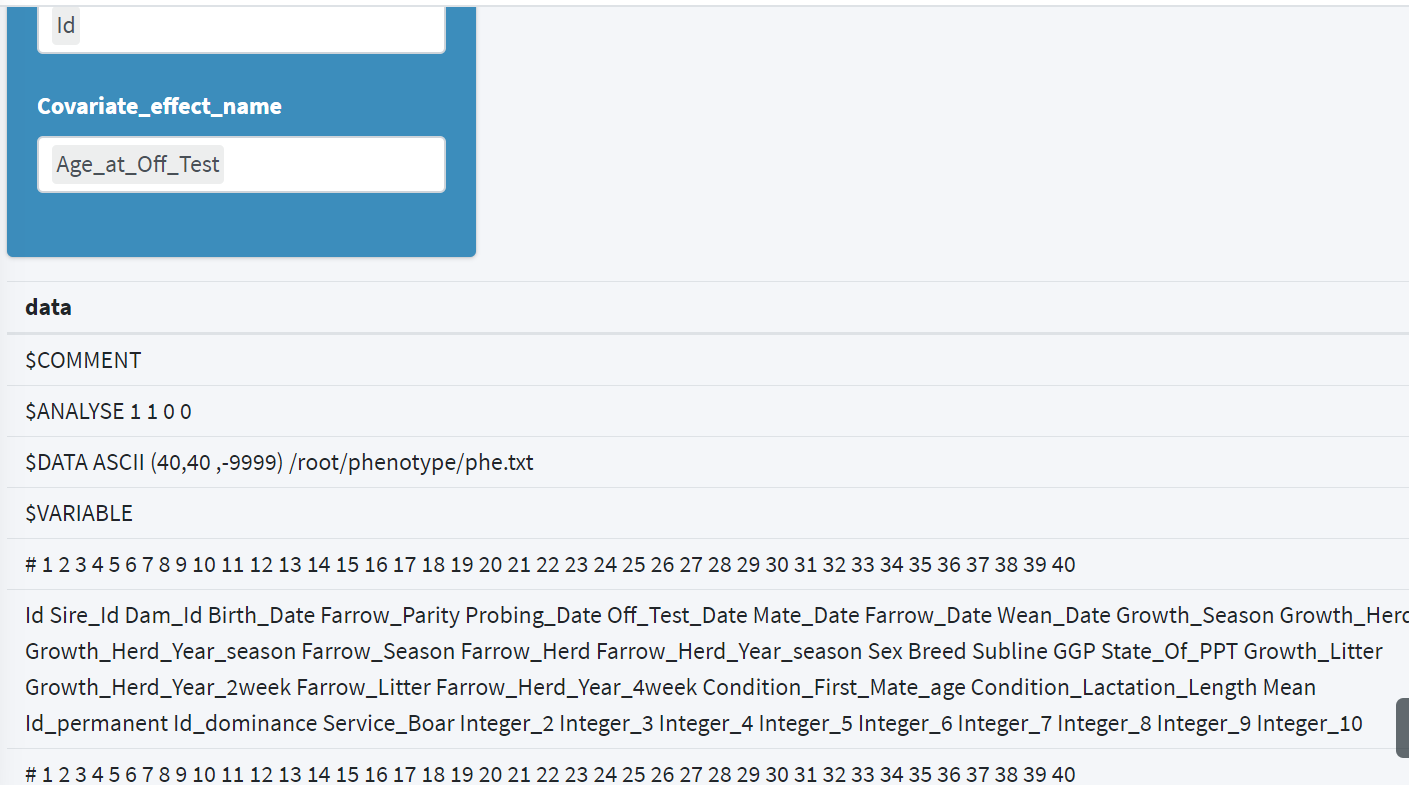
用户可以通过下方的 Download DIR按键即可将生成的 dir 文件下载到本地。
✋SOL界面
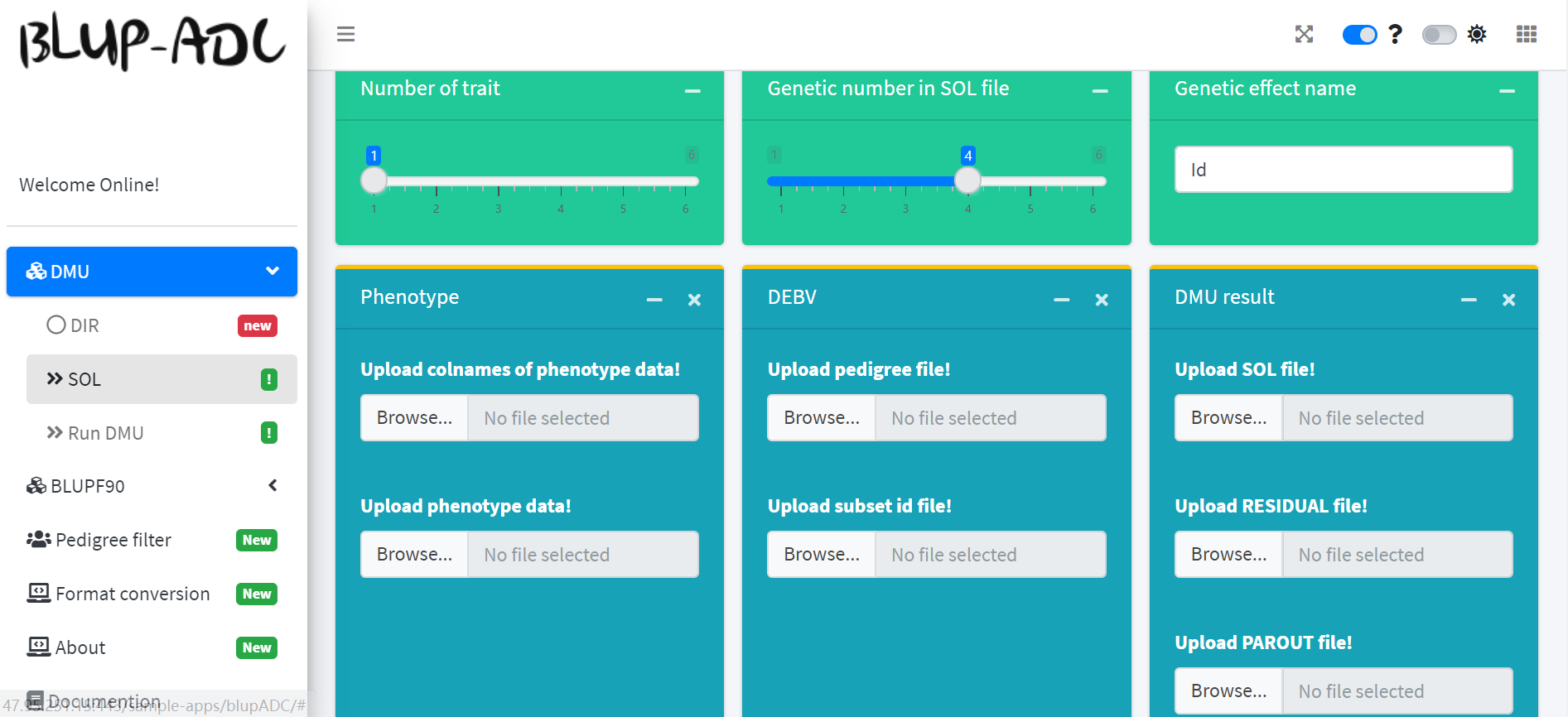
点击左侧的SOL子菜单,我们就能进入SOL界面。
SOL界面的主要功能是整理DMU输出的结果,快速得到EBV、残差、校正表型和DEBV等结果。下面我们来对界面内的各个参数进行逐一讲解:
Number of trait: 要分析的性状的数目Genetic number in SOL file: SOL文件中,遗传效应所对应的数字代码Name of genetic effect: 遗传效应的名称(一般为动植物个体Id)
Phenotype
Upload colnames of phenotype data: 上传表型数据的列名文件。Upload phenotype data: 上传表型数据文件
DEBV(可选:当需要计算DEBV时,才需要上传相应的文件。)
Upload pedigree file: 上传系谱数据文件Upload subset id: 上传需要计算DEBV的个体号数据文件
DMU result
Upload SOL file: 上传DMU输出的SOL文件Upload RESIDUAL file:上传DMU输出的RESIDUAL文件Upload PAROUT file: 上传DMU输出的PAROUT 文件
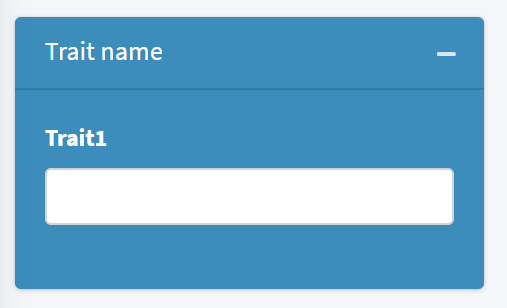
和之前的类似,当我们上传了表型数据的列名文件后,当前界面内同样会新增几个窗口,如下:
当我们输入了对应的性状名称后,界面正下方就会以表格的形式 实时显示 个体对应的EBV, 残差,校正表型和 DEBV,如下:
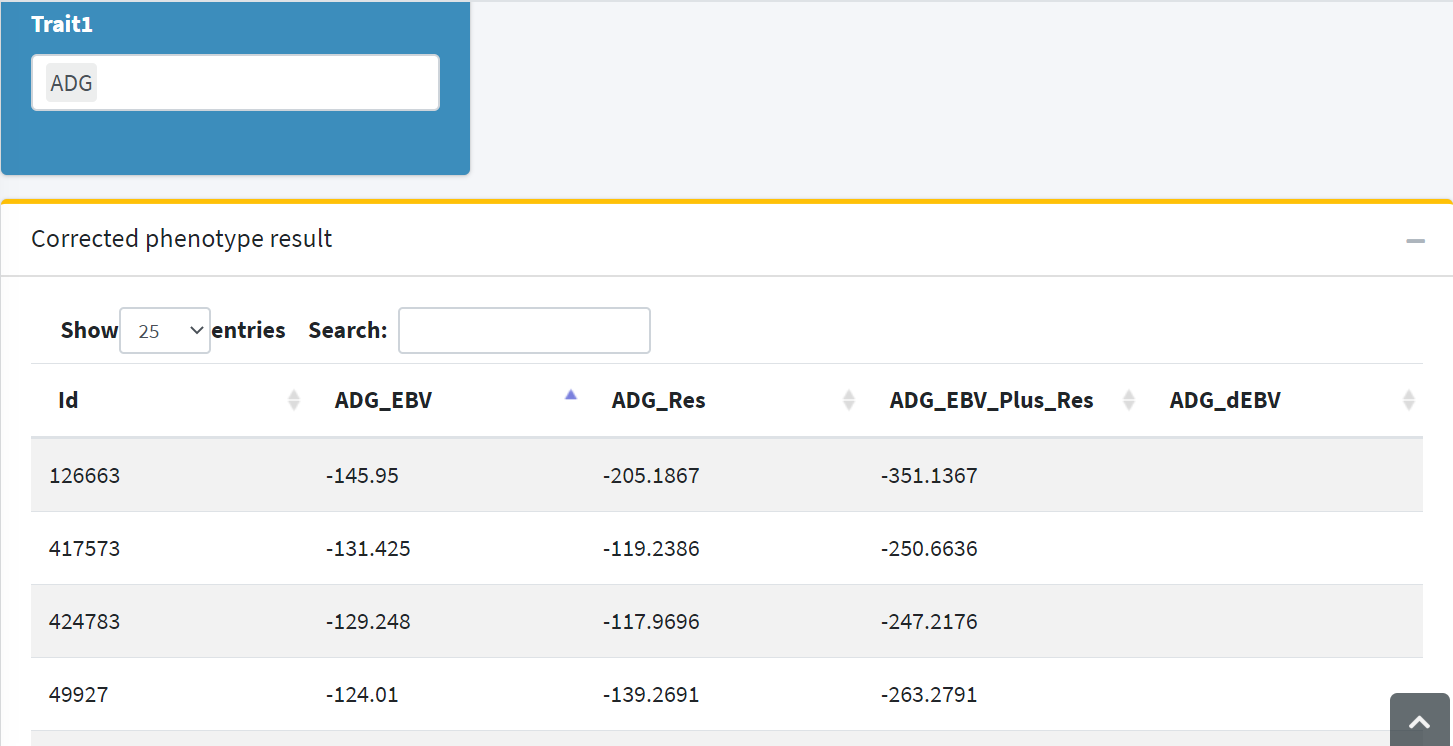
用户可以在表格内轻松的完成查询、排序等操作。
用户可以通过下方的 Download corrected phe按键即可将校正表型文件下载到本地。
✋Run dmu界面
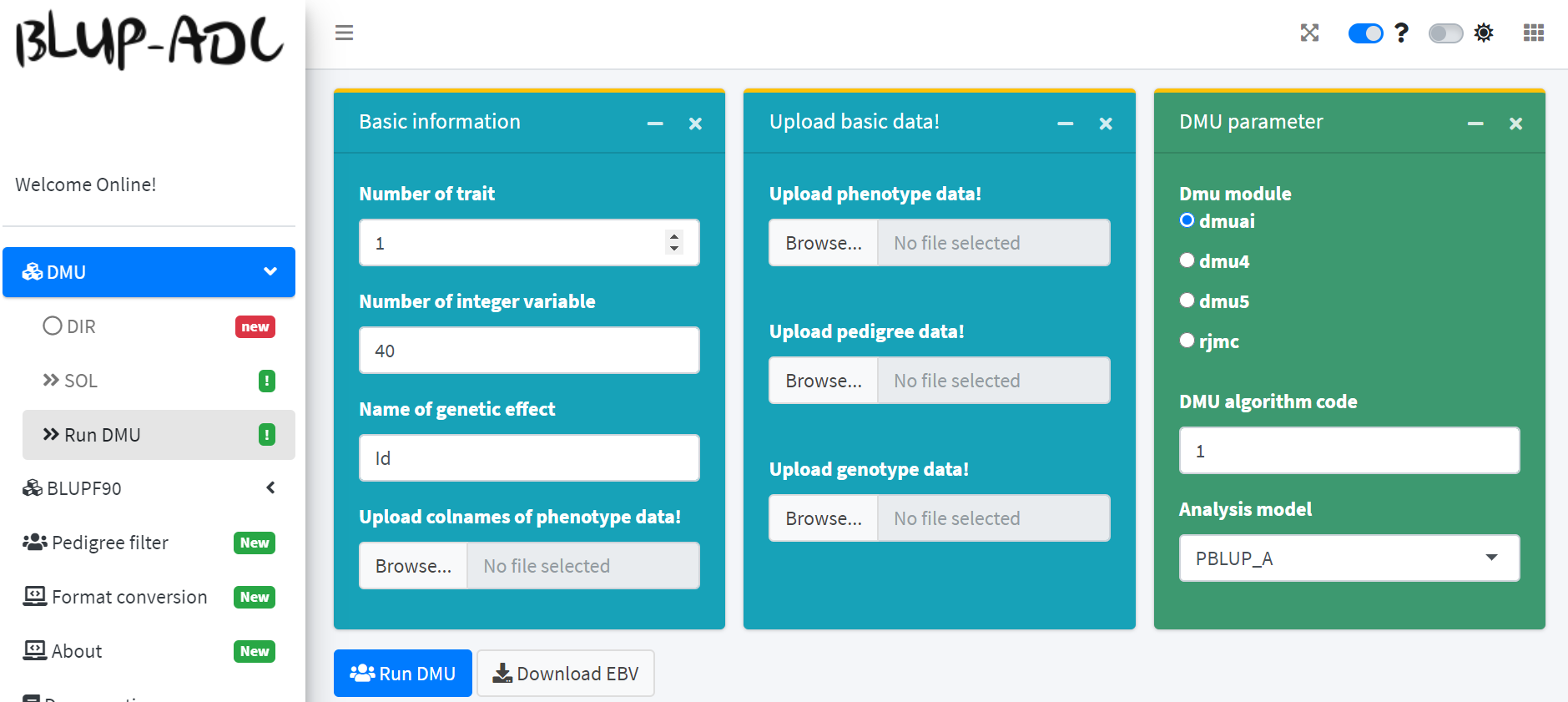
点击左侧的Run DMU子菜单,我们就能进入Run DMU 界面。
Run DMU界面的主要功能是快速帮我们完成DMU相关的分析。其实,Run DMU界面的功能可以看成是 DIR界面和 SOL界面功能的集合体。不过,二者最重要的区别是,Run DMU界面能自动帮我们调用DMU软件并完成相应的分析。
总结起来就是:通过Run DMU 界面,我们只需要提供相关的性状名称和表型、系谱等数据文件后,就能得到最终的个体育种值、残差、校正表型和DEBV等结果。
Run DMU 界面内的各个参数在 DIR界面和SOL界面内都有详细讲解,这里就不再赘述了。
完成了前期的数据准备工作后,用户只需要点击 Run DMU 按键,就能开始DMU的分析了。
由于不同数据量的分析时间是不同的,因此需要耐心的等待。通常情况下,几分钟就能得到结果。结果如下所示:

用户通过点击下方的 Download EBV按键即可将生成的文件保存到本地。